Tutorial para Análise de Dados da COVID-19
Utilizando o R para importação e vizualização de dados da COVID-19

Analisando dados da COVID-19
Como achar uma base de dados para isso?
Muito simples, siga o tutorial que fizemos para vocês e ajude a todos com suas análises.
# install.packages("devtools")
## necessário, pacote de instalação *(certifique-se que todas as suas bibliotecas estão atualizadas)*
library(devtools) #carregamento do pacote
devtools::install_github("Freguglia/datacovidbr")
#instalando o pacote direto do GitHub que contém as pricipais bases de dados
library(datacovidbr) #lendo a biblioteca Para fazer esse exemplar utilizamos a base de dados da brasilio(), mas no pacote que ensinamos vocês a instalar direto do Git, contém outras bases como a do Ministério da Saúde chamada de brMinisterioSaude() entre outras.
Quais os primeiros passos para começar a analisar?
- Quando importamos uma base para dentro do R precisamos antes de tudo conferir nossas colunas para saber exatamente com o que iremos trabalhar.
library(tidyverse) #utilizado para fazer as manipulações dos dados
dados <- brasilio()#atribuindo nossa base de dados para uma variável
head(dados)## # A tibble: 6 x 12
## date state city place_type confirmed deaths order_for_place is_last
## <date> <chr> <chr> <chr> <dbl> <dbl> <dbl> <lgl>
## 1 2020-08-05 AP <NA> state 37318 588 138 TRUE
## 2 2020-08-04 AP <NA> state 36999 581 137 FALSE
## 3 2020-08-03 AP <NA> state 36804 576 136 FALSE
## 4 2020-08-02 AP <NA> state 36639 571 135 FALSE
## 5 2020-08-01 AP <NA> state 36595 568 134 FALSE
## 6 2020-07-31 AP <NA> state 36468 565 133 FALSE
## # … with 4 more variables: estimated_population_2019 <dbl>,
## # city_ibge_code <dbl>, confirmed_per_100k_inhabitants <dbl>,
## # death_rate <dbl>- Mas se o passo anterior não ficou muito claro e não temos o conhecimento de quais são as classes e tipos de variáveis que estamos lidando, usamos a função summary() que nos retorna essas informações entre outras.
summary(dados)## date state city place_type
## Min. :2020-02-25 Length:469924 Length:469924 Length:469924
## 1st Qu.:2020-05-25 Class :character Class :character Class :character
## Median :2020-06-20 Mode :character Mode :character Mode :character
## Mean :2020-06-16
## 3rd Qu.:2020-07-13
## Max. :2020-08-05
##
## confirmed deaths order_for_place is_last
## Min. : 0.0 Min. : 0.00 Min. : 1.00 Mode :logical
## 1st Qu.: 4.0 1st Qu.: 0.00 1st Qu.: 22.00 FALSE:464391
## Median : 17.0 Median : 0.00 Median : 44.00 TRUE :5533
## Mean : 476.2 Mean : 20.06 Mean : 47.97
## 3rd Qu.: 89.0 3rd Qu.: 2.00 3rd Qu.: 70.00
## Max. :585265.0 Max. :24109.00 Max. :161.00
##
## estimated_population_2019 city_ibge_code confirmed_per_100k_inhabitants
## Min. : 781 Min. : 11 Min. : 0.002
## 1st Qu.: 7041 1st Qu.:2500734 1st Qu.: 31.908
## Median : 15510 Median :3136959 Median : 135.100
## Mean : 120141 Mean :3162411 Mean : 402.598
## 3rd Qu.: 34710 3rd Qu.:4107801 3rd Qu.: 468.457
## Max. :45919049 Max. :5300108 Max. :15730.064
## NA's :2147 NA's :2147 NA's :12646
## death_rate
## Min. :0.00000
## 1st Qu.:0.00000
## Median :0.00000
## Mean :0.04561
## 3rd Qu.:0.03930
## Max. :1.00000
## Análise gráfica
Nessa etapa mostraremos dois gráficos cumulativos para os casos de confirmação e mortes da doença no Estado do Paraná. Vale ressaltar que esse material é apenas uma fonte de ajuda para quem gostaria de fazer análises da COVID-19 e não encontra dados ou até mesmo tem dificuldades para isso.
Como analisar graficamente esses dados?
- Como iremos utilizar apenas os dados do estado do Paraná, precisamos fazer algumas modificações para nós mostrar apenas o que queremos, como já carregamos nossa base e um pacote responsável por essas modificações acima em, Quais os primeiros passos para começar a analisar?, vamos dar continuidade com a exploração gráfica.
dados.cidades <- dados%>%filter(dados$state =="PR")
#dentro da coluna state pegamos apenas os dados que forem igual a PR
dados.pr <- dados.cidades%>%filter(dados.cidades$place_type =="state")
#selecionamos dentro da variável dados.cidades apenas os dados que contém o tipo de local
#igual a state
dados.cidades #visualizando os dados## # A tibble: 27,764 x 12
## date state city place_type confirmed deaths order_for_place is_last
## <date> <chr> <chr> <chr> <dbl> <dbl> <dbl> <lgl>
## 1 2020-08-05 PR <NA> state 85317 2164 145 TRUE
## 2 2020-08-04 PR <NA> state 82718 2128 144 FALSE
## 3 2020-08-03 PR <NA> state 80865 2050 143 FALSE
## 4 2020-08-02 PR <NA> state 79605 2005 142 FALSE
## 5 2020-08-01 PR <NA> state 77930 1975 141 FALSE
## 6 2020-07-31 PR <NA> state 76112 1920 140 FALSE
## 7 2020-07-30 PR <NA> state 73488 1861 139 FALSE
## 8 2020-07-29 PR <NA> state 70920 1814 138 FALSE
## 9 2020-07-28 PR <NA> state 69392 1743 137 FALSE
## 10 2020-07-27 PR <NA> state 68001 1703 136 FALSE
## # … with 27,754 more rows, and 4 more variables:
## # estimated_population_2019 <dbl>, city_ibge_code <dbl>,
## # confirmed_per_100k_inhabitants <dbl>, death_rate <dbl>- Utilizaremos o pacote ggplot2 para fazer estes gráficos com uma melhor qualidade
library(ggplot2)#biblioteca para análise gráfica
library(plotly)#biblioteca para análise gráficaGráfico acumulado do número de confirmações de COVID-19 no estado do Paraná
graph_conf <- dados.pr %>%
ggplot(aes(x=date)) +
xlab("Dias/Meses") +
ylab("Confirmados") +
geom_area( aes(y = confirmed), fill = "#35b779", alpha = 0.4) +
geom_line( aes(y = confirmed), color = "#35b779", size = 0.4) +
geom_point(aes(y = confirmed), color = "#35b779", size = 1.3)
graph_conf 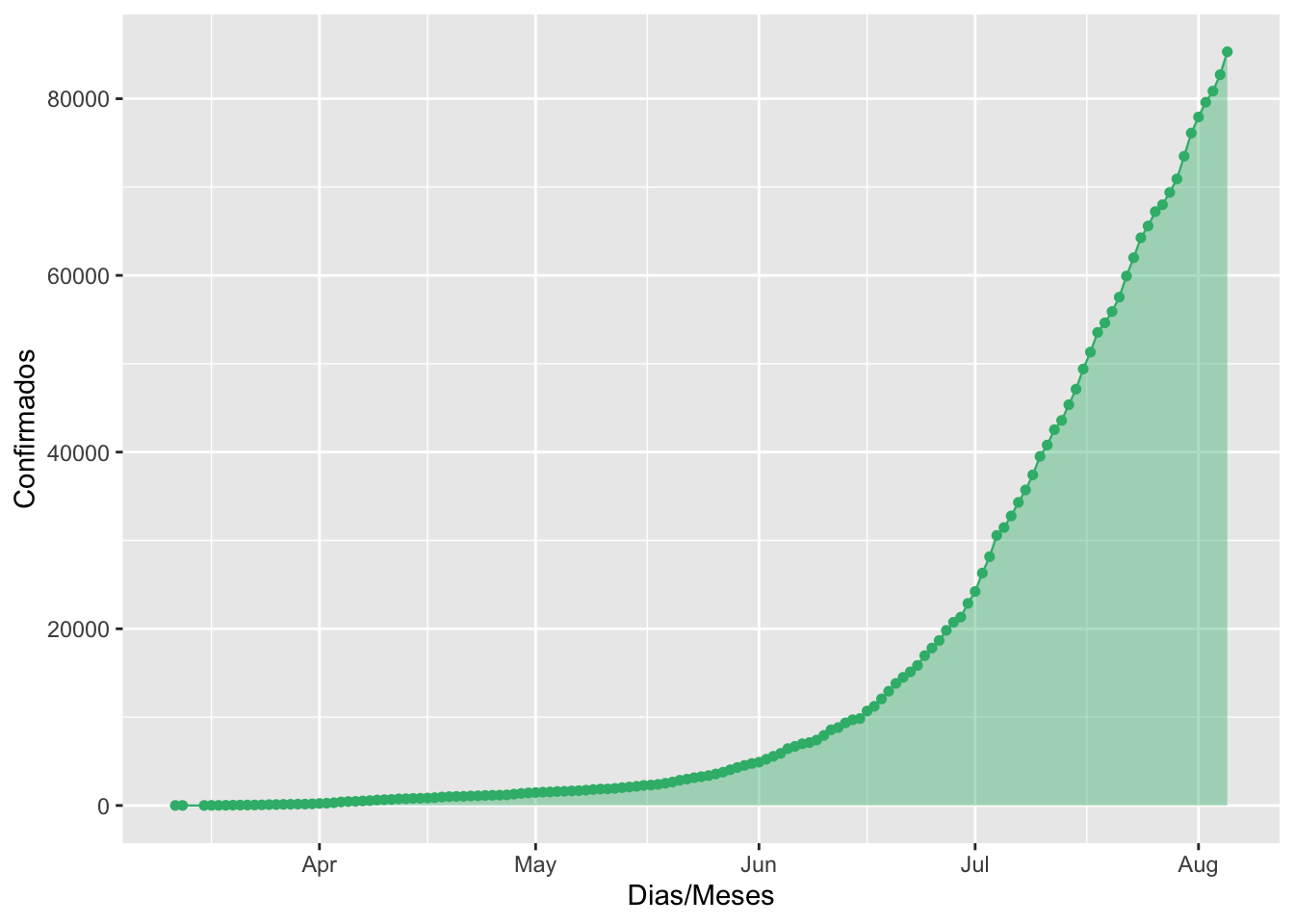
Gráfico acumulado do número de confirmações de óbitos por COVID-19 no estado do Paraná.
graph_deaths <- dados.pr %>%
ggplot(aes(x=date)) +
xlab("Dias/Meses") +
ylab("Mortes") +
geom_area( aes(y = deaths), fill = "#f25a5a", alpha = 0.4) +
geom_line( aes(y = deaths), color = "#f25a5a", size = 0.4) +
geom_point(aes(y = deaths), color = "#f25a5a", size = 1.3)
graph_deaths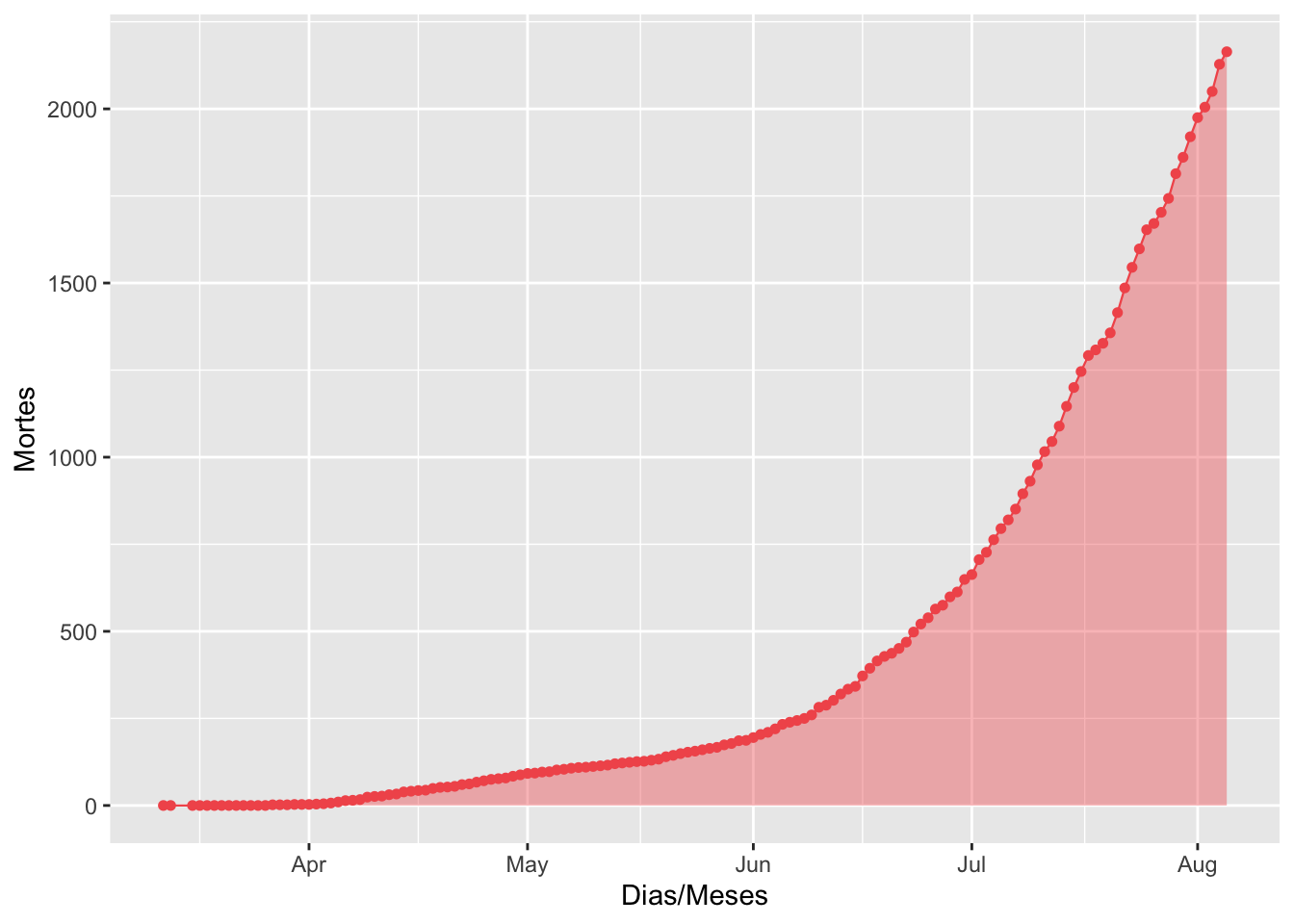
Conscientização de todos
Para concluir este material gostaríamos de pedir a conscientização de todos, saia de casa apenas se for muito necessário e se sair utilize a máscara e higienize suas mãos. Estamos passando por uma situação muito complicada mundialmente, mas se fizermos nossa parte isso acabará rápido e sem maiores problemáticas.